Wo liegt der Schwerpunkt Ihrer Arbeit am Institut für Biotechnologie?
Laura Helleckes: Wir sind beide Doktoranden, die im Bereich der Automatisierung der Entwicklung von Bioproduktionsprozessen forschen. Hintergrund: Für einen solchen Prozess gibt es eine größere Anzahl von Organismen zur Auswahl, und wir können unterschiedliche Betriebsbedingungen schaffen. Für die industrielle Anwendung geht es darum, aus dieser kombinatorischen Vielfalt herauszufinden, welcher Organismus unter welchen Bedingungen am besten für einen bestimmten Produktionsprozess geeignet ist. Am Institut müssen Hunderte dieser automatisch ablaufenden Wachstumsexperimente zeitnah ausgewertet und in Modelle umgesetzt werden. Für diesen Workflow entwickeln wir die nötigen Software-Werkzeuge.
Wie sehen diese Werkzeuge aus?
Michael Osthege: Es sind statistische Werkzeuge. Ich bin Mitglied des Entwicklerteams des Python-Software-Pakets „PyMC3“ für die statistische Modellierung auf Basis sogenannter Bayes‘scher Schätzmethoden, die auch Unsicherheiten einbeziehen. „PyMC3“ ist ein Open-Source-Modellierungstool, mit dem wir am Institut in hohem Durchsatz mehr Informationen aus unseren Experimenten gewinnen können.

Wo liegt der Schwerpunkt Ihrer Arbeit am Institut für Biotechnologie?
Laura Helleckes: Wir sind beide Doktoranden, die im Bereich der Automatisierung der Entwicklung von Bioproduktionsprozessen forschen. Hintergrund: Für einen solchen Prozess gibt es eine größere Anzahl von Organismen zur Auswahl, und wir können unterschiedliche Betriebsbedingungen schaffen. Für die industrielle Anwendung geht es darum, aus dieser kombinatorischen Vielfalt herauszufinden, welcher Organismus unter welchen Bedingungen am besten für einen bestimmten Produktionsprozess geeignet ist. Am Institut müssen Hunderte dieser automatisch ablaufenden Wachstumsexperimente zeitnah ausgewertet und in Modelle umgesetzt werden. Für diesen Workflow entwickeln wir die nötigen Software-Werkzeuge.
Wie sehen diese Werkzeuge aus?
Michael Osthege: Es sind statistische Werkzeuge. Ich bin Mitglied des Entwicklerteams des Python-Software-Pakets „PyMC3“ für die statistische Modellierung auf Basis sogenannter Bayes‘scher Schätzmethoden, die auch Unsicherheiten einbeziehen. „PyMC3“ ist ein Open-Source-Modellierungstool, mit dem wir am Institut in hohem Durchsatz mehr Informationen aus unseren Experimenten gewinnen können.

Wie sieht es mit einer deutschen „rt.live“-Seite aus?
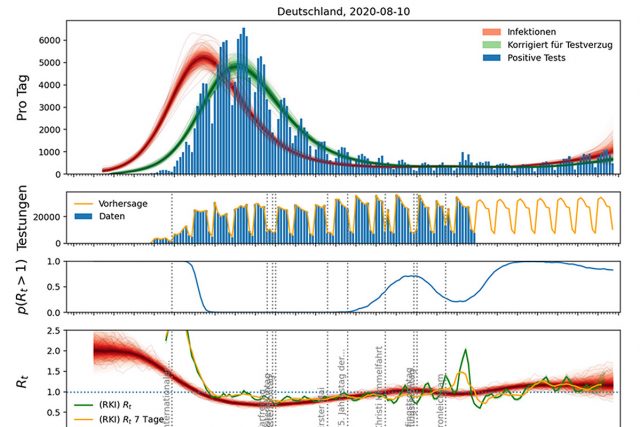
Laura Helleckes: Wir haben aus dem Projekt viel gelernt und untersuchen gerade, in welcher Form das Modell zur Bewältigung einer zweiten Welle in Deutschland genutzt werden könnte. In diesem Zuge stellen wir inzwischen der Öffentlichkeit unsere täglich aktualisierten Vorhersagen unter https://rtlive.de zur Verfügung.
Wo sehen Sie noch Probleme?
Michael Osthege: Anders als bei den Positivergebnissen sind die deutschen Testzahlen nur mit einigen Tagen bis Wochen Verzögerung verfügbar. Wir haben das Modell daher um eine Vorhersage der Gesamt-Testzahlen erweitert. Die Vorhersage wird in der Detail-Ansicht dargestellt. Doch je weiter die letzten Zahlen zurückliegen, desto unzuverlässiger wird sie.
Zur Themenseite Corona-Forschung am Forschungszentrum Jülich